Helsinki Challengen Metabold-tiimin haasteen keskiössä on kysymys siitä, mitä elimistömme pienet molekyylit eli metaboliitit kertovat terveydentilastamme ja miten ne vaikuttavat siihen.
Pienet mutta niin tärkeät metaboliitit
Ihmisen elimistössä on monentyyppisiä pieniä molekyylejä erilaisissa pitoisuuksissa, jopa yli tuhat erityyppistä pientä molekyyliä. Metaboliiteilla on monenlaisia tehtäviä, ne kuljettavat energiaa, ovat solujen rakennuspalikoita, kuljettavat viestejä solujen sisällä ja niiden välillä sekä säätelevät solujen toimintaa.
Kuinka pieni on pieni? Esimerkiksi rypälesokeri eli glukoosi on läpimitaltaan noin nanometrin (metrin miljardiosa), 25000 tuhatta kertaa pienempi kuin ihmisen hiuksen läpimitta ja satoja kertoja pienempi kuin näkyvän valon aallonpituus. Pieniä molekyylejä ei siis voi nähdä eikä muutenkaan helposti havaita suoraan, vaan tarvitaan monimutkaisia mittalaitteita joiden tuottamia signaaleja täytyy vielä käsitellä laskennallisesti. Herkin tunnettu menetelmä pienten molekyylien havaitsemiseksi on massaspektrometria (MS), joka perustuu siihen että eripainoiset ionit voidaan erotella niiden käyttäytymisen perusteella sähkökentässä.
Metaboliittien tunnistamiseksi ei kuitenkaan riitä sen massan mittaaminen, riippumatta mittaustarkkuudesta. Syy on erilaisten vaihtoehtoisten molekyylirakenteiden suuri määrä. Vaikka massan lisäksi tunnettaisiin molekyylikaava, vaihtoehtoisia molekyylirakenteita voi pahimmillaan olla tuhansia. Metaboliittien tunnistusta helpottaa huomattavasti niin sanottu tandemmassaspektrometria (MS/MS), jossa alkuperäinen ioni pilkotaan palasiksi ja palaset erotellaan niiden massan perusteella. Yhdestä molekyylistä saadaan tällöin kokonainen spektri (MS/MS spectrum). Kaksi molekyyliä joilla on sama molekyylikaava, muta eri rakenne, tuottavat usein erilaisen kokoelman palasia, jolloin niiden spektrit näyttäytyvät erilaisina.
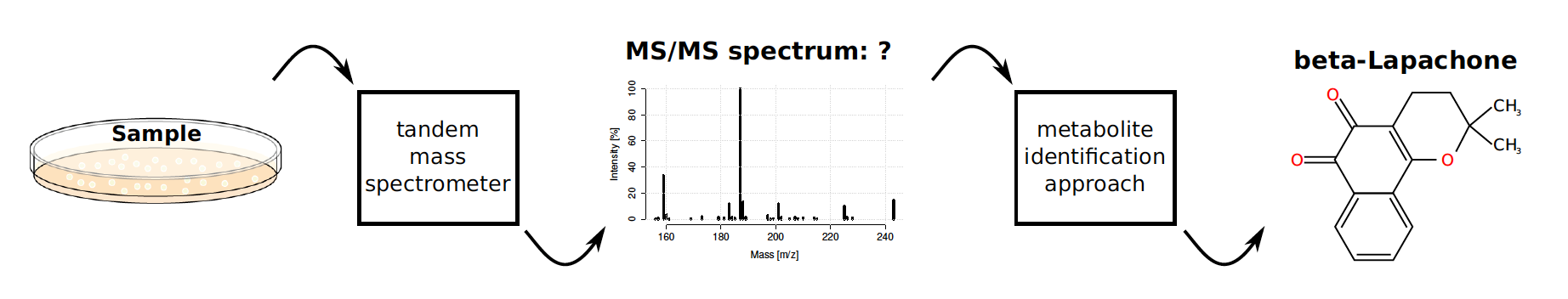
Tietokone apuun datan murskaukseen
Yhden veri- tai sylkinäytteen tuottama mittausdata voi sisältää satoja tandemmassaspektrejä, joista kustakin pitäisi tunnistaa spektriä vastaava molekyylirakenne. Tällaisen aineiston läpikäyminen manuaalisesti on luonnollisesti kallista ja aikaavievää. Tietokoneavusteiset metaboliittien tunnistusmenetelmät ovat onneksi kehittyneet viime vuosina hurjasti. Parhaat ennustemallit pystyvä karsimaan vaihtoehtoisten rakenteiden määrää murto-osaan alkuperäisestä, mikä helpottaa asiantuntijan työtä ja avaa mahdollisuuksia uusiin sovelluksiin.
Aalto-yliopiston KEPACO-tutkimusryhmän kehittämät metaboliittien tunnistusmenetelmät nykyään perustuvat koneoppimiseen. Koneoppimisessa tietokoneelle annetaan tuhansia esimerkkejä, joissa kuvataan tandemmassaspektri ja sitä vastaava oikea molekyylirakenne. Esimerkkien perusteella tietokone muodostaa automaattisesti ennustemallin, joka yhdistää mitatun spektrin ominaisuuksia molekyylien ominaisuuksiin, esimerkiksi sen sisältämiin funktionaalisiin ryhmiin.
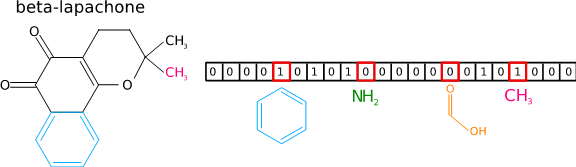
Kun ennustemallille näytetään uusi ennen näkemätön spektri, se ennustaa millaisia ominaisuuksia vastaavalla molekyylirakenteella on. Vertaamalla ennustettuja ominaisuuksia vaihtoehtoisiin molekyylirakenteisiin muodostetaan lopulta järjestetty lista molekyylejä, missä kärkisijoilla ovat molekyylit, joilla sopivat ennustettuihin ominaisuuksiin parhaiten. Tätä lähestymistapaa käyttävät menetelmät voittivat vuonna 2016 kansainvälisen CASMI-kilpailun (http://www.casmi-contest.org) , jossa etsittiin tarkinta metaboliittien tunnistusmenetelmää.
Kohti metabolisen terveyden monitorointia
Metaboliittien tunnistaminen auttaa ymmärtämään, miten liikunta, ravinto, alkoholinkäyttö sekä sairaudet näkyvät metaboliittien tasapainossa. Metabold-tiimin tavoite onkin kehittää metaboliittien monitoroinnista palvelu, jota voivat hyödyntää niin lääkärit, urheilijat, itsensämittajat kuin ’tavalliset’ kuluttajatkin.
Juho Rousu
Kirjoittaja on Aalto-yliopiston professori, jonka tutkimusryhmä (http://research.ics.aalto.fi/kepaco/) kehittää koneoppimismenetelmiä pienille and vähän suuremmillekin molekyyleille. Kuvat: Eric Bach
